- +1
【科技前沿】卜东波/杨怀义/叶盛合作构建基于AI的蛋白质从头设计算法ProDESIGN-LE
蛋白质设计是合成生物学领域当前最难解决的问题之一,同时也是最具潜在价值的问题之一。蛋白质设计的目标是要让设计出来的蛋白质能够具备某种生物学功能,但是实现这一目标同时涉及到了对于蛋白质折叠形式的设计,以及对于蛋白质序列的设计。蛋白质序列设计要能够保证设计得到的氨基酸序列在溶液状态下折叠为目标结构,进而有可能具有目标生物学功能。
传统的蛋白质设计技术多依赖于专家经验,辅以深度突变扫描等筛选技术,成本高,耗时长,且成功率非常低。通过计算技术,尤其是近期发展迅猛的人工智能技术,“理性”设计蛋白质序列,可以大大缩短传统试验和研发的时间,在广阔无垠的蛋白质序列空间中找到符合设计目标的序列,从而创造出更多的具有实用价值的人工蛋白质。
近日,中国科学院计算技术研究所卜东波研究员团队,联合中国科学院微生物研究所杨怀义研究员团队,以及北京航空航天大学医学科学与工程学院叶盛教授团队,共同在Bioinformatics在线发表了题为Accurate and efficient protein sequence design through learning concise local environment of residues的研究论文,发布了一种基于人工智能的蛋白质序列设计算法ProDESIGN-LE,可以快速准确设计与目标结构的主链高度吻合的蛋白质序列。

ProDESIGN-LE的基本思想可以概括成一句话:若每个氨基酸都fit其局部微环境,则整条序列fit整体结构。算法核心是“学习残基的局部微环境,根据每个残基的局部微环境,决定最适合的氨基酸类型”。ProDESIGN-LE从该观点出发,使用邻居和中心残基的相对位置信息刻画局部微环境信息,设计基于Transformer的神经网络来学习残基对其局部微环境的依赖性,然后再利用训练好的网络模型,通过对目标结构的每个位置进行迭代更新获得最终的设计序列,并展现出优秀的精度和效率。
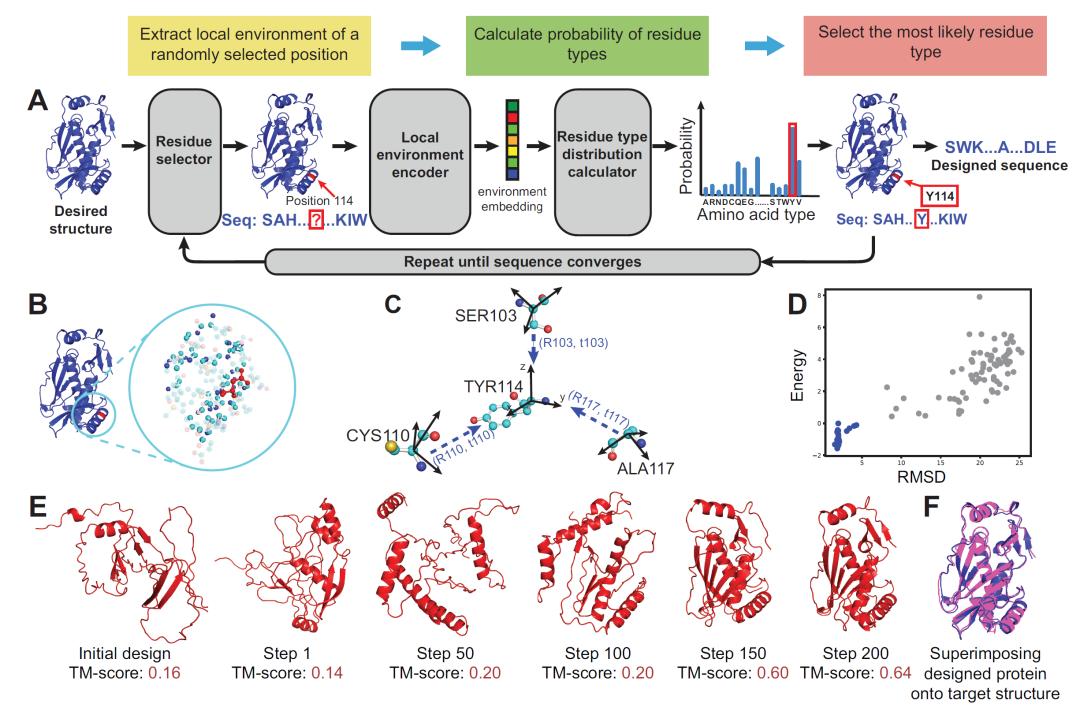
使用Transformer技术根据残基的局部环境信息为其分配适当的残基类型,并通过对序列的每个位置进行迭代更新获得最终的设计序列。在对68个自然蛋白与129个幻觉蛋白的序列设计中,ProDESIGN-LE仅在平均20秒的时间内便可获得预测结构与目标蛋白高度吻合的蛋白质序列。最后,通过为CAT III设计序列并在大肠杆菌中重组表达,作者团队在实验上验证了ProDESIGN-LE的有效性。
以CASP14数据集中提取的68个自然界中出现的蛋白质域为代表,ProDESIGN-LE与3D-CNN,、ProteinSolver、FixBB等广泛使用的蛋白质设计方法比较。ProDESIGN-LE的序列一致性为0.33,其设计序列预测结构的TM-score为0.84,高于其他三个方法并且显著接近天然序列预测结构的TM-score值0.88。同时,通过ProDESIGN-LE设计序列所生成的穿线结构具有更低的能量。此外,消融实验证明ProDESIGN-LE对初始序列并不敏感。
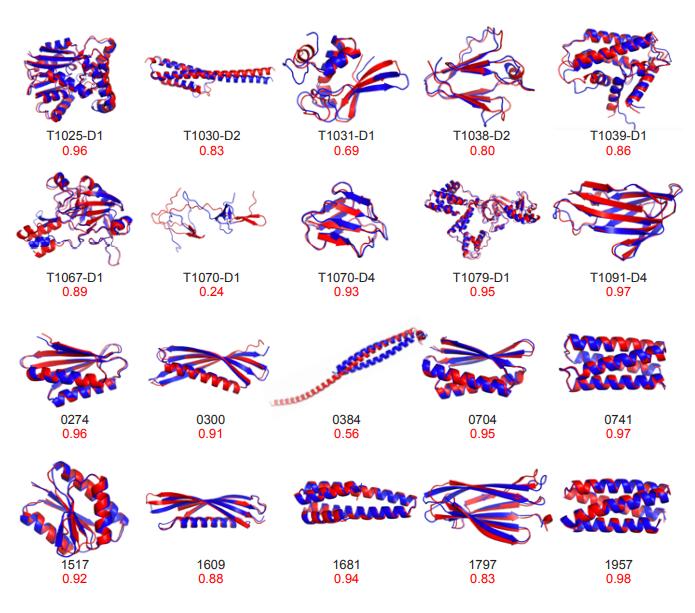
幻觉蛋白来源于倒置的序列到结构的神经网络预测模型的输出结果,是评估蛋白质设计方法普适性的理想测试数据。对于所有129个幻觉蛋白,所有方法产生的设计序列其序列一致性均居于同一水平。在使用ProFOLD-Single或者AlphaFold作为结构预测工具,ProDESIGN-LE设计出的序列,其预测结构的TM-score值均远胜其余模型。此外,穿线结构的平均能量显示ProDESIGN-LE设计的序列更符合幻觉结构。
此外,ProDESIGN-LE在速度和精准度上也表现出了极大的优势。对于前述68种自然蛋白和129种幻觉蛋白,ProDESIGN-LE的平均序列设计速度为20秒。在前10%的局部环境中,ProDESIGN-LE就top-1与top-5的预测准确度分别达到了0.902与0.982。深入研究表明,ProDESIGN-LE预测能力与氨基酸类型密切相关,比如脯氨酸和甘氨酸往往与结构上独特的主链走向有关,因此两者的预测精度较高。
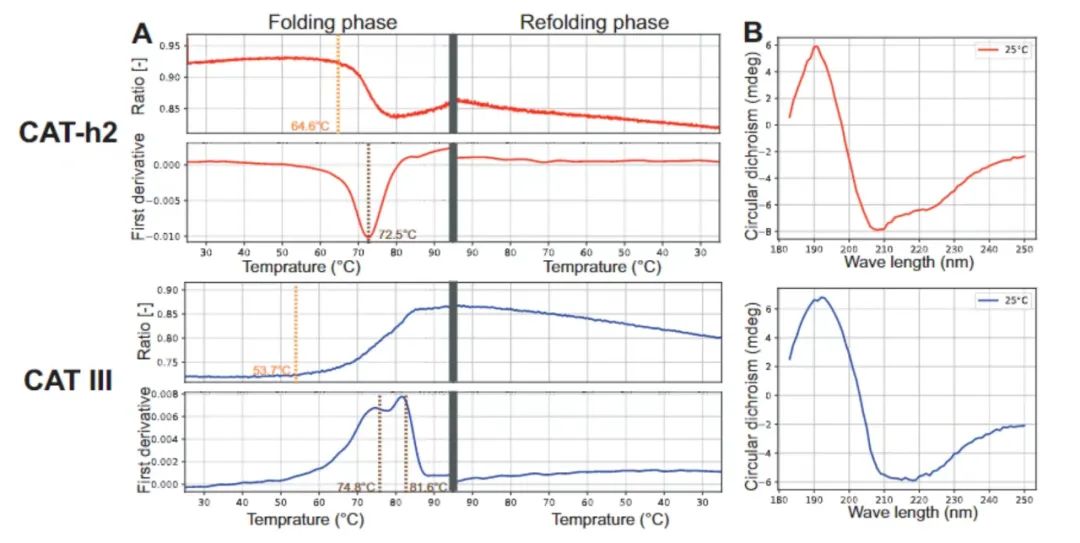
最后,大肠杆菌中用于抵抗氯霉素的CAT III酶被作为设计目标,用以评估ProDESIGN-LE。以其骨架为目标共设计了5种蛋白质序列,并在大肠杆菌中进行了重组表达。实验结果显示,其中3种获得了可溶性表达,并具备了稳定的二级结构。
中国科学院计算技术研究所的卜东波研究员、中国科学院微生物研究所的杨怀义研究员、北京航空航天大学的叶盛教授为论文共同通讯作者,中国科学院计算技术研究所的博士研究生黄斌、中国科学院微生物研究所的范婷文博士、北京航空航天大学的博士研究生王凯悦为共同第一作者。
ProDESIGN-LE源代码已开源(https://github.com/bigict/ProDESIGN-LE),ProDESIGN-LE蛋白质设计服务器可以直接提供开放的设计服务(http://81.70.37.223/)。
原文链接:
https://doi.org/10.1093/bioinformatics/btad122
中国生物物理学会官方订阅号,为BSC会员及生物物理领域专业人士服务。
投稿及授权请联系:bscoffice@bsc.org.cn。
原标题:《【科技前沿】卜东波/杨怀义/叶盛合作构建基于AI的蛋白质从头设计算法ProDESIGN-LE》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。




- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




