- +1
Nature子刊:百图生科宋乐团队开发新型蛋白质结构预测工具,不依赖多序列对比,效率更高
原创 生物世界 生物世界

编译丨王聪
编辑丨王多鱼
排版丨水成文
蛋白质基本上参与了所有的生物过程,在生物体中发挥着至关重要的作用。蛋白质的结构与其在生物过程中的功能高度相关。确定蛋白质结构以了解其功能可以对生命科学做出重大贡献。
近年来,基于人工智能(AI)的蛋白质结构预测技术在预测精度方面取得了长足的进步,在药物和疫苗领域显示出巨大前景。特别是,谷歌旗下公司 DeepMind 开发的 AlphaFold2 将蛋白质结构预测推向了一个新高峰。
主流蛋白质结构预测工具严重依赖于从多序列比对(MSA)中提取的协同进化信息。MSA可以简单地看作是与靶蛋白链序列相似的蛋白质链,MSA与蛋白质序列的协同进化信息有关,这对预测其结构至关重要。然而,过度依赖MSA成为各种蛋白质相关任务的瓶颈,这是因为搜索MSA非常耗时,一个蛋白质需要花费数十分之久,这对于蛋白质设计等任务是破坏性的。
因此,设计一种准确、高效的不依赖MSA的蛋白结构预测方法,有望促进蛋白之研究的发现,从而加速治疗各种疾病的新药的开发。
2023年10月9日,百图生科宋乐博士联合百度自然语言处理部的研究人员,在 Nature 子刊 Nature Machine Intelligence 上发表了题为:A method for multiple-sequence-alignment-free protein structure prediction using a protein language model 的研究论文。
该研究开发了一种名为HelixFold-Single的端到端的蛋白质结构预测方法,该方法结合了大规模蛋白质语言模型(PLM)和AlphaFold2优越的几何学习能力,不依赖多序列比对(MSA),仅从初级结构(氨基酸序列)预测原子三维坐标,从而实现对蛋白质结构的准确预测。此外,HelixFold-Single比目前基于MSA的主流蛋白质结构预测工具(AlphaFold2和RoseTTAFold)耗时少得多,在治疗性蛋白(多肽、抗体和纳米抗体)设计中具有很大的应用潜力。

研究团队认为,大规模的蛋白质语言模型(PLM)可以作为多序列比对(MSA)的替代方案,以学习不依赖MSA的预测的协同进化知识。
基于MSA的方法利用信息检索技术从蛋白质序列数据库中明确捕获目标蛋白质的协同进化信息,而基于PLM的方法在训练过程中将协同进化信息嵌入到大规模模型参数中,并通过模型推理进行隐式检索,其中PLM可视为蛋白质知识库。相比MSA,基于PLM的方法在信息检索方面效率更高。
在过去的几年里,大语言模型(LLM)在自然语言处理领域取得了巨大成功,例如现在爆火的ChatGPT。而现在,已经有一些研究尝试将蛋白质语言模型(PLM)应用于蛋白质结构预测,但预测效果还不尽如人意。
受蛋白质语言模型(PLM)和AlphaFold2进展的启发,研究团队提出了一个端到端不依赖MSA的蛋白质结构预测工具——HelixFold-Single。
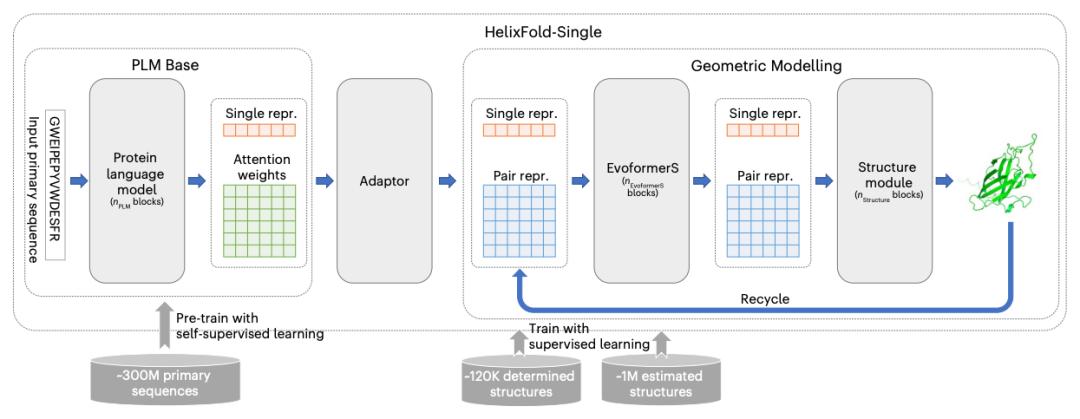
HelixFold-Single中使用的模型由两个主要组件组成——作为基础的大规模蛋白质语言模型(PLM)和来自AlphaFold2的用于蛋白折叠的基本组件。PLM可以将主结构编码为单表征和对表征来学习领域知识。然后集成AlphaFold2中的Evoformer和Structure模块来处理表征,学习几何知识,然后预测原子坐标。这两个组件连接起来,给出一个端到端的可微模型(Differentiable Model)。
HelixFold-Single包含两个训练阶段,在第一阶段,通过掩码语言预测任务,用数百万个未标记的单序列训练大规模PLM。在第二阶段,使用由实验解析和AlphaFold2生成的增强结构组成的蛋白质结构来训练整个模型。
接下来,研究团队将HelixFold-Single与两款主流蛋白质结构预测工具AlphaFold2(由DeepMind公司开发)和RoseTTAFold(由华盛顿大学David Baker实验室开发)在CASP14和CAMEO数据集上进行比较。
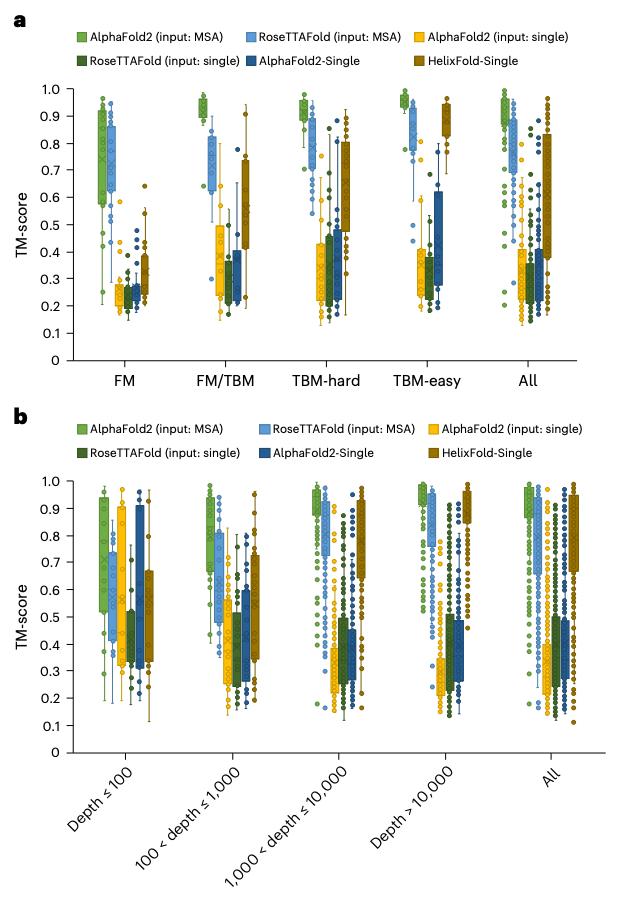
HelixFold-Single在具有足够数量的同源序列的蛋白质上实现了与AlphaFold2和RoseTTAFold相当的准确性。研究团队还分析了HelixFold-Single在具有不同数量同源序列的靶标上的性能:HelixFold-Single能够对大多数靶标,特别是具有大同源家族的靶标提供准确的结构预测。
此外,与基于MSA的的主流蛋白质结构预测工具AlphaFold2和RoseTTAFold方法相比,HelixFold-Single在预测效率上具有很大优势,耗时要少得多,可以应用于需要大量预测的蛋白质相关任务。
具体来说,研究团队探索了HelixFold-Single在各种类型的代表性蛋白质(包括多肽、抗体和纳米抗体)上的预测精度,以评估其在治疗性蛋白质设计中的应用潜力。结果表明,HelixFold-Single在预测这些蛋白质的柔性区域方面表现良好,突出了其在此类应用中的优势。
论文链接:
https://www.nature.com/articles/s42256-023-00721-6
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。




- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




