- +1
eDNA增进北极生物多样性研究:拖网与分子数据的结合分析
【中国绿发会讯】本文约 4400 字,阅读约 8 分钟
环境DNA(eDNA)指的是从环境中采集的DNA样本(可以来自水体、土壤、空气等)。在北极地区的eDNA的应用对于生物多样性研究可以非常有用,因为北极生态系统正在面临气候变化等压力,生物多样性和生态系统结构可能发生变化。通过分析北极海域的eDNA,科学家可以了解该地区的鱼类和海洋哺乳动物多样性,监测物种变化和生态系统健康状况。
近日,“海洋与湿地”(OceanWetlands)小编注意到一项于2024年2月27日在线发表在Progress in Oceanography期刊上的最新研究,来自挪威特罗姆瑟海洋研究所、挪威北极大学渔业科学学院等机构的研究人员探讨了如何利用eDNA来增进对生物多样性的研究,并且与传统的调查方法进行了一番比较。这项研究为了解北极及巴伦支海地区的生物多样性提供了新的视角和数据,对未来的监测和保护工作具有重要意义。
本研究的亮点包括以下几点:一是通过结合eDNA和拖网数据,能够提高检测到的生物多样性水平。其二是建立良好的参考数据库对于准确的分类学分类至关重要,这为分子生态学研究提供了重要支持。另外,研究团队强调:将组合采样作为未来监测北极地区生物多样性的良好策略,特别是巴伦支海的生态系统。

©绿会融媒·海洋与湿地
我们知道,保护自然资源的可持续管理,需要深入了解生态系统的复杂性以及与不同生态系统部分相关的物种。而往往,大部分这方面的知识,是来自于传统的采样方法(例如不同类型的拖网)。然而,随着技术的发展,现在eDNA分析可以提供与传统方法互补的知识。
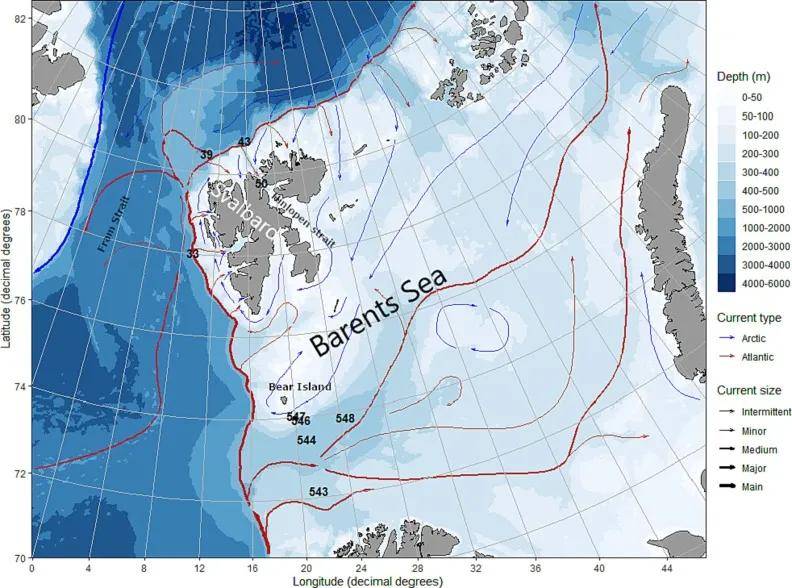
(上图:显示采样位置的地图。具有三位数字的站点来自TIBIA航次(巴伦支海西南部),而具有两位数字的站点来自SI-ARCTIC航次(斯瓦尔巴群岛北部和西部)。图源:Westgaard, Jon-Ivar, et al. )
在这项试点研究中,研究团队从斯瓦尔巴特(Svalbard)北部和西部(NWS)以及巴伦支海(Barents Sea)西南部(SWBS)两个地理区域采集了eDNA样本。他们结合了拖网捕捞、对海洋哺乳动物的目视识别和eDNA采集,对鱼类和海洋哺乳动物的多样性和物种组成进行了一番全面的分析。通过对eDNA样本进行12S MiFish代谢条形码测序,研究团队发现将eDNA数据纳入分析中,可以为研究区域的鱼类和海洋哺乳动物的多样性提供额外的信息。将eDNA数据与拖网数据相结合后,鱼类种类的丰富度从32种增加到了49种。eDNA分析还揭示了两个研究区域之间鱼类群落多样性和组成的显著差异。
(海湿小编注:巴伦支海位于欧洲北部,是挪威和俄罗斯之间的一片海域,位于北极圈内。它是北大西洋中最北的一片海域,与北冰洋相邻,被认为是北极地区的重要海域之一,也是重要的渔业区域。)
该研究强调,传统监测方法仍然主要依赖于针对高商业价值物种的拖网等传统捕捞设备,因此,需要采用补充性方法,来涵盖那些不容易被这些设备捕捉到的物种。结合使用eDNA和传统采样方法,是未来北极及巴伦支海生物多样性监测的良好策略。
研究方法
研究团队在2015年5月(TIBIA)和2016年9月(SI-ARCTIC)进行了两次航次,分别在斯瓦尔巴特北部和西部(NWS)以及巴伦支海西南部(SWBS)的近海地区进行了eDNA和拖网采样。他们在NWS区域的架海沟和SWBS区域的五个站点进行了水样和拖网采样。通过采集海水样本和进行拖网捕捞,科学家们希望探索eDNA在北极海域生物多样性研究中的潜力和优势。
研究团队使用Niskin瓶和CTD测深仪,在不同深度采集了海水样本,并使用0.22微米的Sterivex滤器将海水中的DNA提取出来。这些滤膜在船上冷冻保存,并在后续实验室工作中进行DNA提取和分析。
对于不同地区的海洋生物采样,研究团队使用了不同的拖网装备。对于西南巴伦支海区域(SWBS),他们使用了Campelen拖网,这是一种原本设计用于捕捉虾类的小型底拖网。而在斯瓦尔巴特北部(NWS)地区,他们则使用了Åkra拖网,用于捕捉浮游鱼类。这些拖网采样不仅帮助收集鱼类等生物样本,还有助于了解不同深度和生境下的生物多样性。
此外,研究团队还进行了对海洋哺乳动物的观察。在斯瓦尔巴特北部的航次中,他们通过目视观察记录了海豹、鲸类等海洋哺乳动物的分布和活动情况,为研究生物多样性提供了更全面的数据。
从采集的滤膜样本中,研究团队进行了DNA提取和测序。他们使用了修改后的Qiagen血液和组织提取试剂盒进行DNA提取,并通过PCR扩增12S线粒体基因区域。随后,利用Illumina测序技术对PCR产物进行测序,获取了大量的DNA序列数据。
生物信息学分析是研究中的关键步骤。研究团队使用FASTQ软件检查序列质量,去除引物序列,筛选出符合要求的序列,并对这些序列进行分类学鉴定。他们构建了物种存在/缺失表格,计算了各个样本的物种丰富度和群落组成,通过统计学方法探索不同地点和深度之间的生物多样性差异。
研究结论和讨论
该研究通过测序技术对水域样本进行了分子生态学分析,结果显示了来自北极地区海洋生物多样性的重要发现。首先,研究对测序库进行了质量过滤,最终获得了10,178,857条序列,这些序列被归类为875个分子操作分类单元(MOTUs)。在这些MOTUs中,有59个MOTUs拥有超过10个reads,其中37个MOTUs被归类为“鱼类”,另外8个被归类为“海洋哺乳动物”。
在进一步分析之前,研究还移除了14个MOTUs,其中包括陆生哺乳动物和鸟类。37个鱼类MOTUs中,有23个能够根据97%相似性标准归类至物种水平,而所有8个海洋哺乳动物MOTUs也能够按照相同的标准归类至物种水平。研究还移除了那些已知分布范围超出研究区域的MOTUs,因为这些物种通常是太平洋物种,缺乏大西洋的参考序列。
研究发现,在所有样本中共检测到了六个物种,其中五种属于商业上重要的物种,包括大西洋真鳕(Gadus morhua)、深水红鱼(Sebastes mentella)、黑线鳕(Melanogrammus aeglefinus)、马舌鲽(Reinhardtius hippoglossoides)和大西洋鲱(Clupea harengus)。
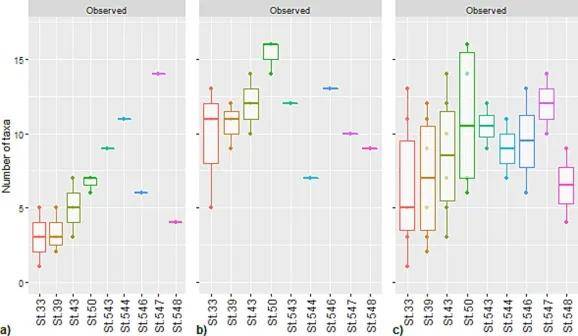
(上图:每个采样站点观察到的鱼类物种数量,分别为(a)拖网数据,(b)eDNA数据,(c)eDNA和拖网数据的组合。图源:Westgaard, Jon-Ivar, et al. )
这些结果为北极地区海洋生物多样性提供了重要的基础数据,为未来的生态研究和保护工作提供了有价值的参考和依据。
通过使用环境 DNA(eDNA)和拖网两种方法进行调查,研究发现,与传统拖网相比,eDNA 是一种更有效的工具,可以检测到更多的物种,并揭示了一些拖网无法捕获的 "隐藏" 物种。
研究结果显示,eDNA检测到的物种数量比拖网显著更高,尤其在斯瓦尔巴群岛地区,eDNA数据使物种数量几乎翻了一番。eDNA方法能够检测到传统方法难以捕获的物种,如小型、深海或难以接近的物种。尽管eDNA数据不包含个体生活史信息,但它可以确认斯瓦尔巴群岛和巴伦支海存在哪些鱼类、以及海洋哺乳动物物种。
研究还观察到了物种组成的空间差异,显示了一些物种在两个地理区域内和之间的异质性。洋流被认为是影响海洋环境中eDNA分布的重要因素,它可以将颗粒(即eDNA)输送到很远的距离,使不同区域的eDNA信号趋于一致。此外,稀释效应和降解也会影响eDNA信号的可检测距离。
关于鱼类的垂直分布,eDNA数据显示在垂直水柱中没有明显的物种结构差异,这与拖网数据形成对比。这可能是由于eDNA颗粒的平均下沉速度较慢,使得在释放的深度层颗粒停留时间较长。
比较eDNA和拖网数据显示,将eDNA纳入传统的拖网调查可以显著增加检测到的分类单元数量。
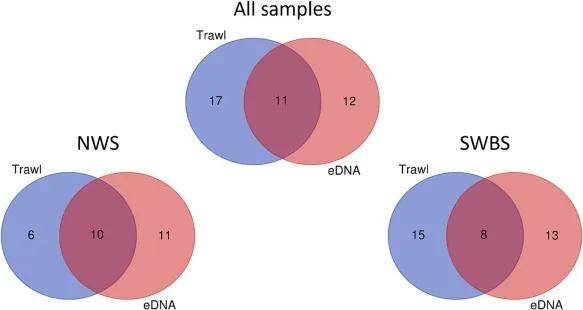
(上图. 比较eDNA和拖网数据结果的鱼类分类单元的维恩图(NWS:斯瓦尔巴群岛西北部,SWBS:巴伦支海西南部)。图源:Westgaard, Jon-Ivar, et al. )
在海洋哺乳动物方面,视觉观察和eDNA方法的一致性表明,eDNA可以作为研究海洋哺乳动物的有效补充采样工具。作者团队尤其指出,通过目视观察和环境DNA(eDNA)方法发现,使用12S元条形码技术(12S metabarcoding)可以有效地收集跨越多个营养级海洋哺乳动物数据。甚至在没有观察员登船的情况下,eDNA也能为巴伦支海的海洋哺乳动物提供观察数据。但是,由于对环境因素如何影响研究中eDNA的空间和时间持久性了解甚少,所以需要借助来自其他地方的eDNA数据进行解释。
未来改进方面,该研究建议:使用多个标记物覆盖更广泛的分类范围,并建立精心策划的本地参考数据库,以提高eDNA数据集的分类分辨率。
这项研究明确指出,将eDNA采样与传统拖网调查相结合,对于了解北极地区鱼类和海洋哺乳动物的物种多样性至关重要。通过结合来自eDNA和拖网采样区域的数据,可以增加对物种丰富度的检测。为了提高eDNA数据集的分类分辨率,尚需进一步努力,重点是建立精心策划的本地参考数据库,使得这种数据更具实用性。研究团队主张,未来监测北极地区生物多样性(包括巴伦支海)的一个良好策略是采用联合采样方法。
感兴趣的“海洋与湿地”读者可以参看这项研究的全文,参见:
Westgaard J I, Præbel K, Arneberg P, et al. Towards eDNA informed biodiversity studies–Comparing water derived molecular taxa with traditional survey methods[J]. Progress in Oceanography, 2024, 222: 103230.
https://doi.org/10.1016/j.pocean.2024.103230
海洋与湿地·小百科
分子操作分类单元
分子操作分类单元(Molecular Operational Taxonomic Units,简称MOTU),是一种基于分子数据对生物进行分类的方法。它通过对DNA或RNA序列进行聚类分析来划分MOTU,每个MOTU代表一群具有高度相似性(通常>97%)的序列。MOTU在许多情况下可以作为传统分类单元的补充或替代。
MOTU的概念最初是由Schloss等人在2011年提出的,旨在解决传统分类方法中的一些局限性。因为传统分类方法主要基于形态学特征来划分物种,但形态特征可能存在可塑性,并且难以用于识别微生物等难以观察的生物。相比之下,MOTU方法基于分子数据,具有更高的客观性和可重复性。MOTU方法在微生物学、生态学、进化生物学等领域有着广泛的应用,比如可以用于分析环境样本中的微生物群落组成、鉴定新的或未描述的物种、研究种群遗传学和进化关系、监测物种的分布和丰度,等等。随着分子数据的不断积累,MOTU方法也在一步步发展和完善中。
分子生态学
分子生态学(molecular ecology)是一门综合了分子生物学和生态学原理的学科,开始于1950年代,旨在研究生物体与环境之间的相互作用和生态系统的结构与功能。它是与许多学科前沿都有交集的前沿学科,通过分析生物体内的分子组成和遗传信息,探索生物多样性、物种分布、群落组成及其响应环境变化的机制。分子生态学的研究方法包括DNA测序、遗传标记、基因表达分析等,这些方法可以帮助科学家们深入了解生物体的生态角色、适应性和进化历史,从而促进对生态系统的保护和管理。在本文中提到的这项研究中,研究人员利用分子生态学方法,特别是环境DNA(eDNA)技术,来研究北极地区的鱼类和海洋哺乳动物多样性。
12S元条形码技术
12S元条形码技术(12S metabarcoding)是一种基于DNA元条形码的分子生物学技术,用于鉴定和分析生物多样性。它通过扩增和测序生物体中的12S rRNA基因片段来实现。12S rRNA基因是核糖体RNA的一部分,存在于所有真核生物中,并且具有高度保守的区域和可变区域。通过比较不同物种12S rRNA基因的序列差异,可以对物种进行鉴定和分类。
事实上,12S元条形码技术的原理是利用12S rRNA基因的保守区域设计通用的引物,对环境样品中的DNA进行扩增。扩增产物经过高通量测序后,可以获得大量的12S rRNA基因序列。通过将这些序列与已知的12S rRNA基因序列数据库进行比对,就可以鉴定出样品中存在的物种。本文中在“海洋哺乳动物”部分,强调了12S元条形码技术可以有效地收集跨越多个营养级海洋哺乳动物数据。
END
本文仅代表资讯,不代表平台观点。
欢迎转发(请注明来源)。
新闻源 | 海洋学进展
编译 | 王芊佳
审核 | Sara
排版 | Sara
本文来自“中国绿发会”微信公众号
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。




- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




